近日,福建海峡科技研究院(未来技术学院)基因组学中心秦源教授团队完成题为“Chromosome-scalegenessequenceofSuaedaglaucashedslightonsalterstressleranceinhalophytes”的研究论文农林大学,已在园艺学领域顶级期刊《园艺研究》上发表论文。

土壤盐分 日益引起全球农作物生产和人类可持续发展的关注。因此,了解耐盐机制并识别耐盐基因以增强作物对盐胁迫的耐受性至关重要。S. glauca是一种很好适应海水环境的盐生植物,具有在细胞内吸收和保留高盐浓度的独特能力,特别是在其叶子中,这表明存在独特的耐盐机制。
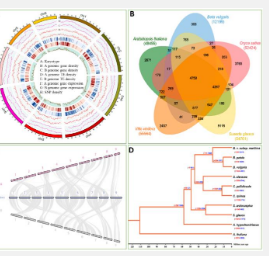
在这项研究中,作者 对S. glauca基因组进行了从头测序 。基因组大小为 1.02 Gb(由两组单倍型组成),包含 54,761 个带注释的基因,包括等位基因和重复序列。比较基因组分析揭示了S. glauca 和B. vulgaris的基因组之间存在很强的同线性。在S. glauca 基因组中,70.56% 包含重复序列,其中 Retroelements 最为丰富(图 1)。
利用S. glauca基因组的等位基因感知组装 ,我们研究了分析样本中的全基因组等位基因特异性表达。结果表明,启动子序列的多样性可能有助于一致的等位基因特异性表达(详细信息请参阅全文)。典型的被子植物花由四个轮生:萼片、花瓣、雄蕊和心皮,尽管某些物种可能会出现这种排列方式的偏差。在 S. glauca中,仅观察到三个轮生,第一轮显着退化,第二轮表现出萼片状特征。第三轮和第四轮显示正常表型(图2A)。确认青萼第二轮的萼状身份 花,我们分析了参与光合作用和叶绿素合成相关途径的基因的表达。聚类热图显示,在第二轮中检查的基因的表达模式与萼片中的相似。此外,对ABCE基因家族的系统分析揭示了青花草花形态的形成,表明A类基因的功能障碍是青花草没有花瓣的原因 (图2)。
为了 从基因组角度深入了解碱蓬属植物的耐盐机制,我们对碱蓬属 植物进行了比较基因组分析,包括碱蓬(苋 科碱蓬 )和其他非碱蓬属植物。 -来自同一科的耐盐物种。结果显示,这两种耐盐碱蓬 物种的基因家族显着扩展,总共包含 1840 个基因(图 3A)。对这些基因的富集分析揭示了与DNA 修复、染色体稳定性、DNA 去甲基化相关的基因本体 (GO) 术语的显着富集、 阳离子结合和红/远红光信号通路在碱蓬物种 的共享扩展基因家族中 (图3B)。与 苋科非盐生物种相比,碱蓬物种 的扩展基因 主要富集于“ DNA/染色质稳定性维持”功能。这表明“DNA/染色质稳定性维持”的作用可能在碱蓬植物的耐盐性中发挥着至关重要的作用 。
为了进一步验证上述观察结果,我们对盐处理的碱蓬植物进行了时间 RNA 序列分析。盐处理后不同时间点差异表达基因的聚类分析如图4A、B所示。值得注意的是,过渡基因被认为是青冈应对盐胁迫的稳定响应机制 。有趣的是,叶片中转变上调的基因主要与DNA修复和染色体稳定性有关,以及与脂质生物合成过程 和类异戊二烯代谢过程相关的GO富集。 这些结果与之前对S. glauca的分析一致 基因家族扩展(图4C)。此外,转录因子的全基因组分析表明 FAR1 基因家族显着扩展(详细信息请参阅全文)。然而,还需要进一步研究来确定 FAR1 基因家族在 S. glauca 耐盐性中的确切作用。
在这项研究中,我们从头组装了S 的单倍体基因组。 染色体水平上的 青光眼。基因组序列质量高,注释广泛,可作为盐生植物青冈的参考基因组。此外,我们阐明了盐生植物中涉及“DNA/染色质稳定性维持”的新型耐盐机制。据我们所知,这种耐盐机制是盐生植物最近发现的,可能为增强作物耐盐性提供新的见解和途径。
标签: